(approfondimento 6 di Potenziale genetico e condizioni di processo nella determinazione della forza del glutine, della digeribilità e dell’immunogenicità)
Nel glutine (soprattutto gliadine e, in parte, glutenine) esiste una forte sovrapposizione tra:
-
resistenza alla digestione gastrointestinale
-
potenziale immunogenico (soprattutto nella celiachia)
ma i due concetti non sono equivalenti: la resistenza è spesso una condizione favorente, mentre l’immunogenicità richiede anche specifiche regole di riconoscimento immunologico.
1) Perché molte sequenze immunogeniche sono anche resistenti
Le regioni più “problematiche” del glutine sono ricche di prolina (P) e glutammina (Q). Questo profilo:
-
ostacola il taglio da parte delle principali proteasi umane (pepsina, tripsina, chimotripsina), che hanno bassa capacità di scindere vicino alla prolina;
-
favorisce la persistenza di oligopeptidi lunghi (10–30+ aa) nel lume intestinale.
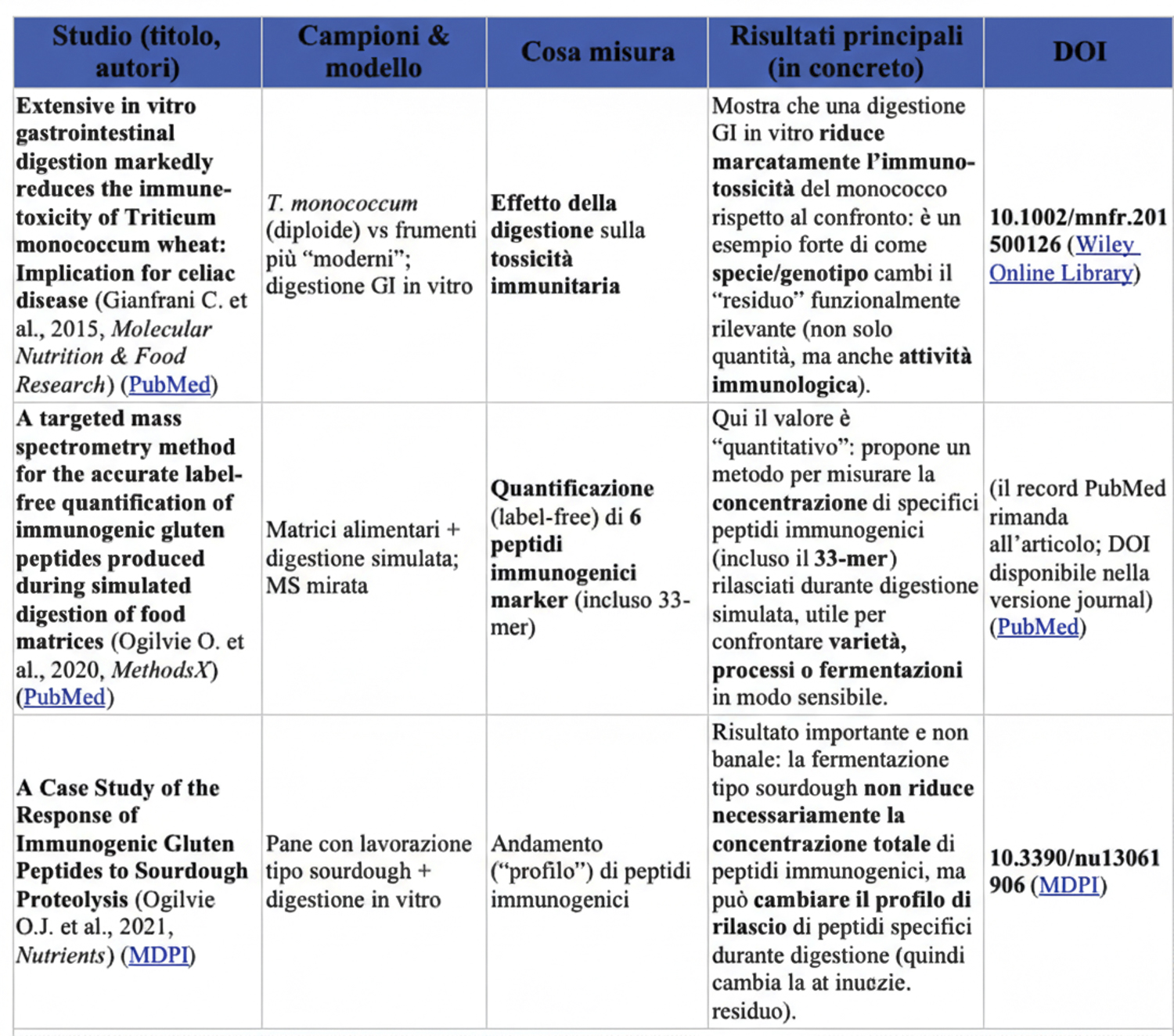
Questo punto è descritto bene in review e studi sperimentali sulla digestione del glutine e sulla persistenza di peptidi come il 33-mer. (Cambridge University Press & Assessment)
2) Perché la resistenza aumenta la probabilità di “rimanere immunogenici” dopo digestione
Un peptide che resiste:
-
resta abbastanza lungo da contenere epitopi completi (o più epitopi sovrapposti);
-
può generare, con tagli parziali, sotto-frammenti che conservano ancora sequenze riconoscibili.
In altre parole: non è solo “sopravvivere” alla digestione, ma sopravvivere mantenendo motivi di sequenza compatibili con la presentazione immunitaria.
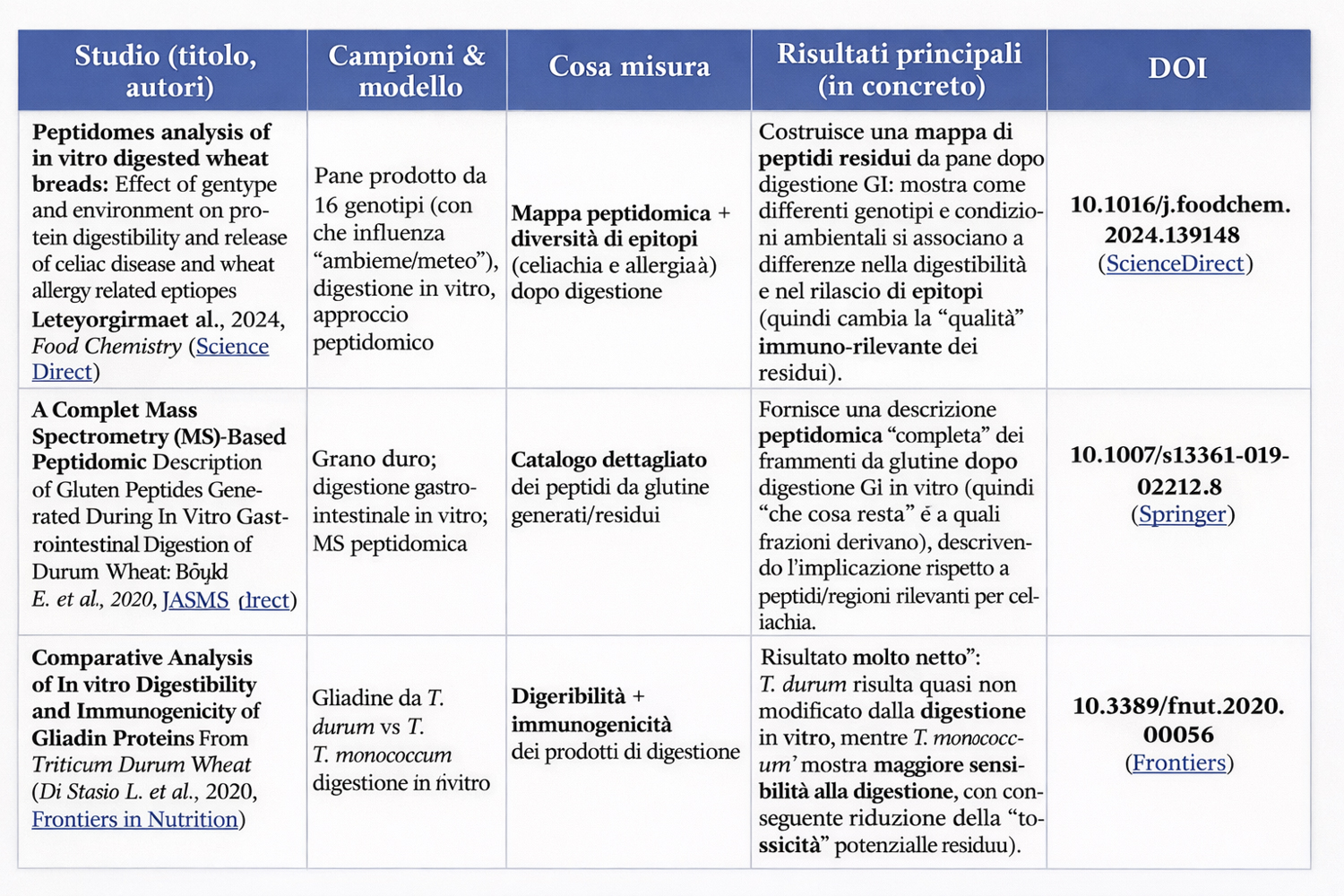
Studi di peptidomica/digestione in vitro su prodotti di frumento mostrano che il profilo di peptidi residui include spesso regioni note per epitope-densità e resistenza. (ScienceDirect)
3) Cosa rende un peptide davvero immunogenico (oltre alla resistenza)
Per innescare la risposta T nella celiachia, un peptide deve:
-
essere presentabile da HLA-DQ2/DQ8 (vincoli di sequenza e “ancore”);
-
spesso diventare più affine tramite deamidazione da parte della transglutaminasi tissutale (TG2) (conversione di Q→E in contesti specifici);
-
essere riconosciuto da T-cellule specifiche.
Quindi è possibile avere peptidi molto resistenti che però:
-
non legano bene HLA-DQ2/DQ8,
-
non sono buoni substrati per TG2,
-
e/o non corrispondono a epitopi T noti.
Un riferimento classico sulla presentazione HLA-DQ2 di peptidi del glutine è disponibile su PNAS. (pnas.org)
4) Esempio concreto: peptide resistente ma non immunogenico
Un esempio molto utile (anche se ingegnerizzato) è descritto da Bethune et al.: gli autori hanno creato analoghi del 33-mer in cui alcune glutammine chiave sono sostituite (es. NNN-33-mer e HHH-33-mer). Questi analoghi:
-
restano resistenti alla digestione simulata (pepsina e anche digestione duodenale con proteasi pancreatiche/brush border),
-
ma non sono apprezzabilmente riconosciuti da TG2, HLA-DQ2 o T-cellule specifiche della celiachia.
Questo dimostra sperimentalmente che resistenza alla digestione ≠ immunogenicità, anche quando la lunghezza e la “prolina-ricchezza” restano simili. (PMC)
Nota: è un esempio “pulito” perché mantiene la caratteristica di resistenza ma spezza (con modifiche mirate) i requisiti immunologici di riconoscimento.
5) Sintesi
Le sequenze immunogeniche del glutine tendono a essere sovrarappresentate tra i frammenti resistenti alla digestione, perché la resistenza consente la persistenza di peptidi sufficientemente lunghi e ricchi di epitopi; tuttavia l’immunogenicità richiede anche compatibilità con la presentazione HLA-DQ2/DQ8 e spesso la modificazione (deamidazione) mediata da TG2.
Approfondimento
Finora non è stata esplorata in modo sistematico e profondo la variabilità genetica e tecnologica di tutto il pool di frammenti resistenti alla digestione, perché la maggior parte degli studi si concentra sui peptidi immunogenici noti piuttosto che sul repertorio completo dei frammenti proteolisi-resistenti in relazione a genotipo/processo. (Frontiers)